Haut de page
Chaque jour, les sciences analytiques génèrent un volume considérable de données. Pour faire face à cette situation, de nouvelles approches sont indispensables afin de collecter, analyser et stocker ces importantes quantités de données. L’Institut des Sciences Analytiques, avec la plateforme de Chimiométrie et de Modélisation CHEMOD, dispose d’un cluster de calcul haute performance (HPC) qui intègre des serveurs de calcul et d’analyse de données massives, permettant ainsi de gérer efficacement le traitement des données. Le cluster est composé, à ce jour, de 30 nœuds de calcul dédiés à la Chimiométrie, aux Machine Learning et à l’Intelligence Artificielle. Grâce à cet outil, nous mettons à la disposition des chercheurs et des entreprises des outils puissants pour la modélisation numérique, l’analyse de données omiques (LC-MS, GC-MS, DI-MS, RMN, etc.), l’analyse de données spectroscopiques (IR, RAMAN, RAMAN imager, etc.), la modélisation QSAR/QSPR…
Équipements
Les projets financés:
La plateforme collabore sur différents types de projets académiques (ANR, IHU, projets européens …) et industrielles. Voici le détail de quelques projets académiques dans lesquelles la plateforme CheMod est impliquée :
- ANR NESOREACH (2009-2013): Nouveaux solvants « éco-compatibles » de substitution pour le respect de REACH – NEw « eco-friendly » SOlvents for REACH substitution – NESOREACH
- ANR ACTIPHEN (2014-2018): ACTIPHEN vise à évaluer le potentiel d’extraits de végétaux contenant des phénols antimicrobiens de 150 à 3 000 g.mol-1 de masse moléculaire pour la conservation des aliments. Ce choix est notamment justifié par les observations suivantes :l’activité antimicrobienne des phénols présents dans les extraits végétaux a été évaluée de façon moins systématique que leur activité antioxydante, les extraits végétaux antimicrobiens les plus étudiés sont les huiles essentielles
- ANR PLAN-TOX (2019-2023): Approches Protéomique et Lipidomique pour la compréhension des mécanismes moléculaires de toxicité en lien avec l’altération du métabolisme lipidique chez l’espèce sentinelle Gammarus fossarum durant le cycle de reproduction – PLAN-TOX.
- IDEX de Lyon WANTED(2019-2022): Which Atmospheric Nanoparticles Trigger Heatlh Diseases? , le projet WANTED a pour objectif de caractériser la composition chimique de la fine fraction de ces aérosols organiques secondaires, et de faire un lien avec leur toxicité sur différents systèmes cellulaires et cliniques
- Projet FEDER COVIDAIR (2020): L’étude clinique COVIDAir évalue une nouvelle méthode pour détecter la Covid-19 via la caractérisation des molécules présentes dans l’air expiré.
- Projet PHRC-N HOPE-SPLIT (2022-2025): Split ex-vivo lors de la perfusion hypothermique oxygénée du greffon en transplantation adulte et pédiatrique : Essai randomisé multicentrique
- Projet IHU EVEREST (2023-2033): L’IHU Everest, porté par l’Institut d’Hépatologie de Lyon, vise à relever les défis actuels et futurs de l’hépatologie, et plus particulièrement contribuer à la guérison des maladies chroniques du foie grâce à une médecine de précision et au développement de thérapies innovantes.
Les équipements de la plateforme CheMod:

La plateforme CheMod possède un cluster de calcul du type HPC dédié aux diverses applications de la Chimiométrie (plans d’expériences, Analyses de données, Machine Learning) aux sciences analytiques. Ce cluster est composé, à ce jour, de 30 nœuds de calculs : chaque nœud de calcul possède au moins 256G de RAM et au moins 20 cœurs (CPU).
Galerie de résultats
Mots clés
Chimiométrie, Plans d’expériences, Analyses multivariées, analyses d’images, Machine Learning
Personnels
Chercheurs et enseignants-chercheurs
Soutien à la recherche
Publications (sources HAL)
Articles dans une revue
- Thomas Alexandre Brunet, Yohann Clément, Valentina Calabrese, Jérôme Lemoine, Olivier Geffard, et al.. Concomitant investigation of crustacean amphipods lipidome and metabolome during the molting cycle by Zeno SWATH data-independent acquisition coupled with electron activated dissociation and machine learning. Analytica Chimica Acta, 2024, 1304, pp.342533. ?10.1016/j.aca.2024.342533?. ?hal-04535105?
- Valentina Calabrese, Thomas Alexandre Brunet, Davide Degli-Esposti, Arnaud Chaumot, Olivier Geffard, et al.. Electron-activated dissociation (EAD) for the complementary annotation of metabolites and lipids through data-dependent acquisition analysis and feature-based molecular networking, applied to the sentinel amphipod Gammarus fossarum. Analytical and Bioanalytical Chemistry, 2024, ?10.1007/s00216-024-05232-w?. ?hal-04507595?
- Guillaume Rossignol, Xavier Muller, Thomas Alexandre Brunet, Valeska Bidault, Valerie Hervieu, et al.. Comprehensive bile acid pool analysis during ex-vivo liver perfusion in a porcine model of ischemia-reperfusion injury. Scientific Reports, 2024, 14 (1), pp.2384. ?10.1038/s41598-024-52504-7?. ?hal-04425302?
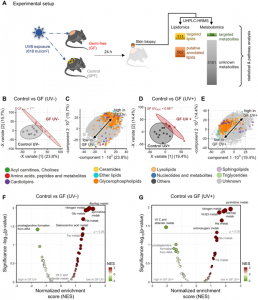
- Vijaykumar Patra, Natalie Bordag, Yohann Clément, Harald Köfeler, Jean-Francois Nicolas, et al.. Ultraviolet exposure regulates skin metabolome based on the microbiome. Scientific Reports, 2023, 13 (1), pp.7207. ?10.1038/s41598-023-34073-3?. ?hal-04091111?
- Pierre Lanteri, yohann clement, Jérôme Randon. Une situation pédagogique sur microcontrôleur pour faire émerger les besoins en analyse de données - le tri des matières plastiques. L'Actualité Chimique, 2023, 487, pp.37. ?hal-04240537?
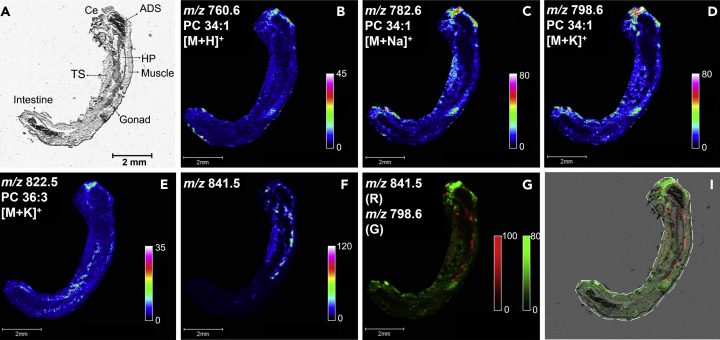
- Julien Faugere, Thomas Alexandre Brunet, Yohann Clément, Anabelle Espeyte, Olivier Geffard, et al.. Development of a multi-omics extraction method for ecotoxicology: investigation of the reproductive cycle of Gammarus fossarum. Talanta, 2023, 253, pp.123806. ?10.1016/j.talanta.2022.123806?. ?hal-03773623?
- Valentina Calabrese, Arnaud Salvador, Yohann Clément, Thomas Alexandre Brunet, Anabelle Espeyte, et al.. Challenges and perspectives in MS-based omics approaches for ecotoxicology studies: An insight on Gammarids sentinel amphipods. Frontiers in Analytical Science, 2023, 3, pp.1118494. ?10.3389/frans.2023.1118494?. ?hal-03986050?